U-net网络
U-net网络:医学图像分割的里程碑
1. 简介
U-net是一种卷积神经网络架构,最早由Olaf Ronneberger、Philipp Fischer和Thomas Brox在2015年提出,用于生物医学图像分割。该网络因其U形结构而得名,已成为医学图像分割领域的基准模型。U-net在训练数据有限的情况下也能获得精确的分割结果,这使其在医学影像分析中特别有价值。
2. 网络结构
U-net的架构由两部分组成:收缩路径(编码器)和扩展路径(解码器),整体结构呈”U”形。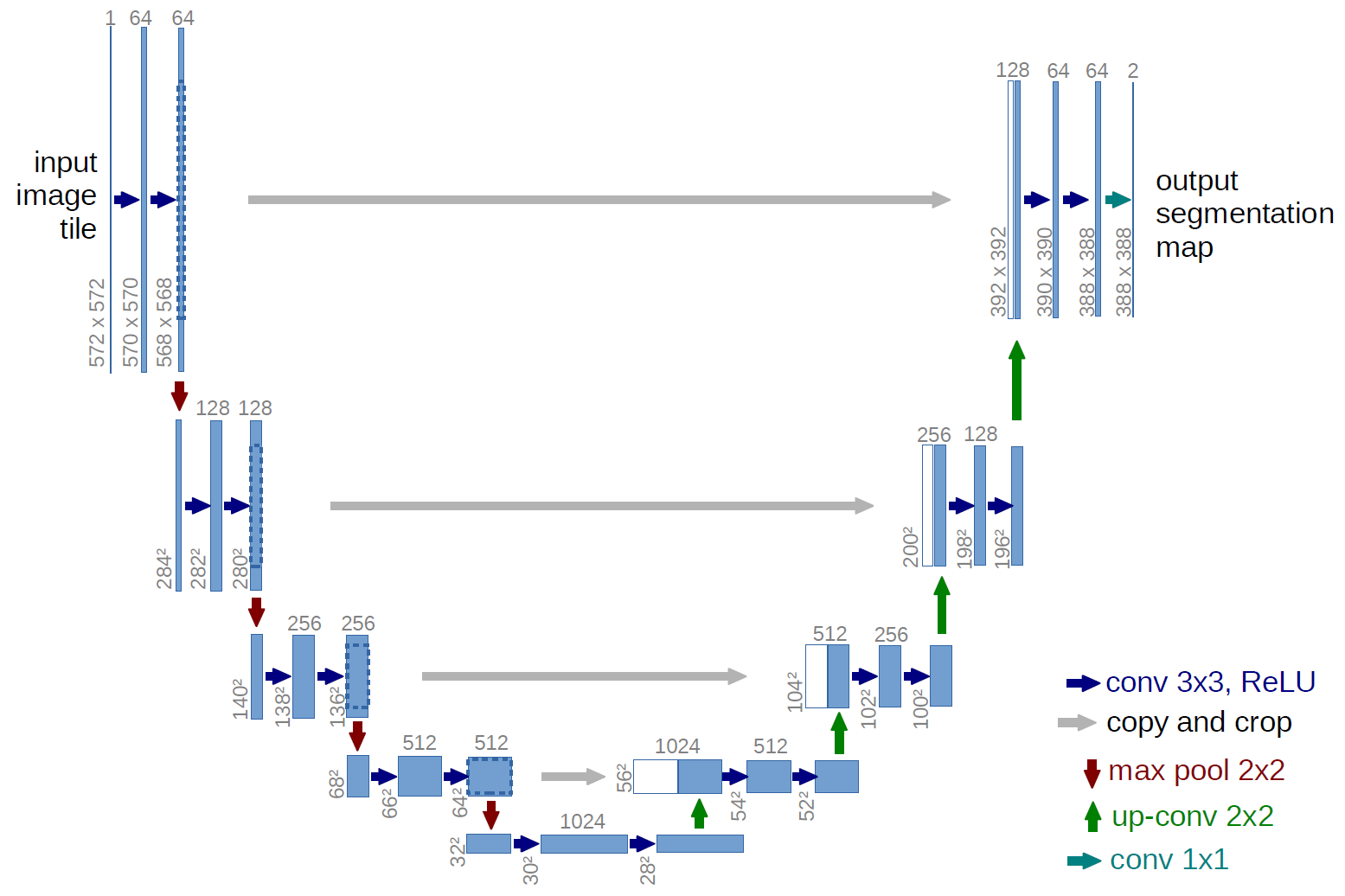
2.1 收缩路径(下采样)
收缩路径遵循典型卷积网络的架构,从左到右依次为:
- 初始层:输入图像首先经过两次3×3卷积操作,每次卷积后接批归一化和ReLU激活函数
- 下采样块:共4个下采样块,每个包含:
- 2×2最大池化(步长为2)进行下采样,将特征图尺寸减半
- 两次3×3卷积(无填充),每次卷积后接批归一化和ReLU
- 每次下采样后特征通道数翻倍(64→128→256→512→1024)
- 底部:位于网络最深处,包含两个3×3卷积层和ReLU激活函数,特征通道数为1024
2.2 扩展路径(上采样)
扩展路径包含4个上采样块,每个块包含:
- 上采样操作:使用2×2转置卷积(步长为2)进行上采样,将特征图尺寸扩大一倍
- 特征融合:将上采样的特征与收缩路径对应层的特征图进行拼接(跳跃连接)
- 这些跳跃连接对于恢复分割过程中丢失的空间信息至关重要
- 特征处理:拼接后进行两次3×3卷积操作,每次后接批归一化和ReLU
- 特征减半:每次上采样后特征通道数减半(1024→512→256→128→64)
2.3 最终输出层
- 网络最后使用1×1卷积层将特征图映射到所需的类别数量(C个通道)
- 对于二分类任务,通常使用sigmoid激活函数
- 对于多分类任务,使用softmax激活函数
- 输出特征图尺寸与输入图像相同,实现像素级的精确分割
2.4 尺寸变化说明
假设输入图像为572×572:
- 每次3×3卷积(无填充)会使特征图尺寸减少4个像素
- 四次下采样后,特征图尺寸变为28×28
- 上采样过程中恢复尺寸,最终输出为388×388
- 这种设计使网络能够对边界区域进行更精确的分割
3. U-net的创新点
U-net相比传统CNN有几个关键创新:
- 对称的U形结构:使网络能够同时捕获上下文和精确定位
- 跳跃连接:将编码器的特征直接传递给解码器,保留空间信息
- 无全连接层:保留空间信息,可用于任意尺寸的输入
- 数据增强策略:通过弹性形变等技术有效扩充训练样本
4. 应用场景
U-net在医学图像领域有广泛应用:
- 细胞分割
- 器官及病变区域分割
- 血管分割
- 肿瘤检测
- 眼底图像分析
5. U-net变种
随着技术发展,U-net架构衍生出多个变种:
- 3D U-net:扩展到三维数据处理
- V-net:用于3D医学图像分割,使用残差连接
- Attention U-net:引入注意力机制改善特征选择
- TransUnet:结合Transformer和U-net的优势
6. 代码实现
以下是使用PyTorch实现U-net的简化版本:
1 | |
7. 训练技巧
成功训练U-net网络的几个关键点:
- 数据增强:旋转、缩放、弹性变形等
- 权重初始化:使用适当的初始化方法(如He初始化)
- 损失函数选择:常用Dice损失或交叉熵与Dice损失的组合
- 学习率策略:通常采用衰减学习率
- 批量归一化:帮助加速训练和提高稳定性
8. 优缺点分析
优点
- 在小数据集上表现优异
- 结构简单,训练效率高
- 可处理任意大小的输入
- 分割精度高,特别适合医学图像
缺点
- 对全局上下文信息获取有限
- 池化操作可能导致细节信息损失
- 在类别不平衡数据上需要特殊处理
- 原始结构计算量较大
9. 结论
U-net凭借其简洁而强大的设计,已成为医学图像分割任务的基础架构。虽然已有许多改进版本,但其核心思想—结合多尺度特征和保留空间信息的能力—仍然是现代图像分割网络的基石。随着深度学习技术的不断发展,U-net及其变种将继续在医学图像分析领域发挥重要作用。
10. 参考资料
- Ronneberger O, Fischer P, Brox T. U-net: Convolutional networks for biomedical image segmentation. MICCAI 2015.
- Zhou Z, et al. UNet++: A nested U-Net architecture for medical image segmentation. DLMIA 2018.
- Chen L C, et al. DeepLab: Semantic image segmentation with deep convolutional nets, atrous convolution, and fully connected CRFs. IEEE TPAMI 2017.
- Isensee F, et al. nnU-Net: a self-configuring method for deep learning-based biomedical image segmentation. Nature Methods 2021.
U-net网络
http://neutrino.top/2025/03/28/U-net网络/